9月24日,国际学术期刊Genome Biology在线发表了中国科学院上海营养与健康研究所生物医学大数据中心张国庆研究员与南方科技大学生命科学学院系统生物学系王泽峰讲席教授团队的合作论文“A foundation language model to decipher diverse regulation of RNAs”。该研究构建了一种基于深度神经网络的预训练模型,可微调预测pre-mRNA的剪接位点、mRNA的翻译效率、mRNA的降解率和内部核糖体进入位点(Internal Ribosome Entry Site,IRES)等多个RNA调控相关的下游任务,揭示了RNA中调控元件的序列特征并鉴定新型翻译调控元件,为理解RNA调控机制和优化RNA的生物医学应用提供了新工具和新思路。
在真核生物中,RNA转录、剪接、翻译和降解等生物学过程受到顺式调控元件、RNA结构和反式作用因子的严格调控。解析RNA的多层次调控对于研究基因表达分子机制和设计RNA药物具有重要意义。然而由于调控的复杂和数据量的不足,目前构建RNA调控的预测模型仍然面临挑战。
为了突破上述瓶颈,研究团队设计并训练了基于多层transformer编码器架构的RNA语言模型LAMAR。研究首先下载处理约1500万条哺乳动物和病毒的基因和转录本序列,通过掩码学习进行无监督预训练,预先提取RNA的序列特征;之后使用含有标签的数据集微调模型,实现RNA调控高效预测。
研究测试了LAMAR模型在多个下游任务中的性能。其中,LAMAR模型在mRNA翻译效率和降解率预测任务中分别取得0.66和0.65的Spearman相关系数指标,相比最优基线模型提升7%和8%。另外,LAMAR模型在剪接位点预测任务中取得0.96的PR-AUC指标,与最优基线模型SpliceAI的性能相当。
研究还使用公开数据集微调模型预测病毒和真核IRES,取得0.985的AUROC指标。研究进一步预测RNA病毒基因组中潜在的新IRES,并在多个细胞系中测试其中305条序列驱动环形RNA翻译的效率。研究发现序列的预测概率与翻译活性呈正相关,提示模型模拟筛选新型调控元件的能力。
目前,LAMAR模型已上传至Github(https://github.com/rnasys/LAMAR),供科研人员预测pre-mRNA的剪接位点、mRNA翻译效率、降解率和IRES,或使用自己的数据集微调模型。
中国科学院上海营养与健康研究所张国庆研究员、南方科技大学生命科学学院王泽峰教授、美国北卡罗莱纳大学教堂山分校胡玥博士后为论文共同通讯作者。中国科学院上海营养与健康研究所博士研究生周翰文、美国北卡罗莱纳大学教堂山分校胡玥博士后为论文共同第一作者。该研究得到了科技部国家重点研发计划、国家自然科学基金、中国科学院战略性先导科技专项(B类)、上海市科技创新行动计划、上海市市级科技重大专项等项目的资助。
论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-025-03752-x
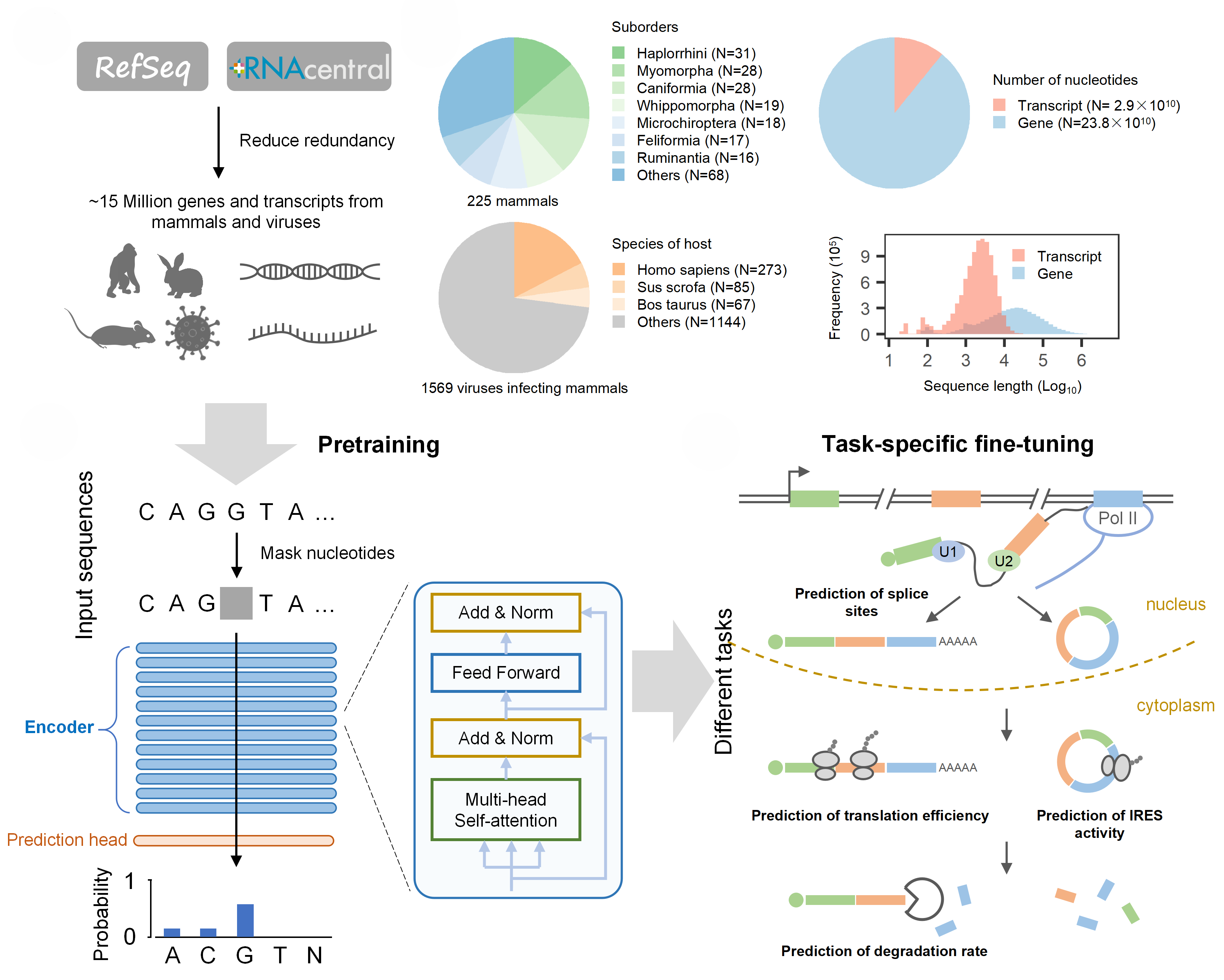
图1:LAMAR模型架构及研究流程图:模型首先使用大量基因和转录本序列进行无监督预训练,再使用含有标签的数据集进行微调解决RNA调控下游任务。
推送单元:生物医学大数据中心、科技规划与任务处